Οδηγός χρήσης libRadtran.
2025-02-19
| Αρχείο pdf |
| Η εργασία αυτή διανέμεται υπό την άδεια: |
| Creative Commons - Αναφορά Δημιουργού - |
| Μη Εμπορική Χρήση - Παρόμοια Διανομή 4.0 Διεθνές. |
| http://creativecommons.org/licenses/by-nc-sa/4.0/ |
| Για διορθώσεις και προσθήκες επικοινωνήστε με Thanasisn github.com/thanasisn. |
Σημείωση: Ο πηγαίος κώδικας (source code) που παρατίθεται εδώ είναι ένα παράδειγμα εφαρμογής κάποιων τεχνικών. Σε περίπτωση που θέλετε να τον χρησιμοποιήσετε, προτείνουμε να δοκιμαστεί η καταλληλότητα του για την επιθυμητή χρήση, πρώτα σε μη σημαντικές εφαρμογές (non-critical). Τα παραδείγματα κώδικα εδώ, διαφέρουν από αυτά που χρησιμοποιούμε, λόγω της συνεχής εξέλιξης και βελτίωσης κατά την χρήση τους.
Δημιουργία εκτελέσιμου (build/compile) της Libradtran 2.0 σε GNU/Linux.
Το παράδειγμα αυτό έχει εφαρμοστεί σε λειτουργικό σύστημα Debian και βασίζεται στις οδηγίες που βρίσκονται στις ιστοσελίδες www.libradtran.org/doku.php?id=download και www.libradtran.org/doc/INSTALL.
Εγκατάσταση βιβλιοθηκών (libraries) συστήματος.
Εκτός από τα εργαλεία που περιγράφονται στις οδηγίες, είναι αναγκαίες και κάποιες άλλες βιβλιοθήκες για να μπορεί να γίνει compile του εκτελέσιμου uvspec. Η εγκατάστασή τους μπορεί να γίνει με την εντολή:
sudo apt-get install libgsl0* netcdf-* libnetcdf*Εγκατάσταση του uvspec στο σύστημα.
Το εκτελέσιμο που δημιουργήθηκε μπορεί να γίνει διαθέσιμο στο σύνολο του συστήματος βάζοντας ένα link της θέσης του στο φάκελο των αρχείων συστήματος.
sudo ln -s '/path/to/libRadtran-2.0-64/bin/uvspec' '/usr/bin/uvspec'Το βήμα αυτό δεν είναι απαραίτητο, αλλά διευκολύνει την εκτέλεση του αρχείου από οποιοδήποτε σημείο του συστήματος χωρίς την ανάγκη να χρησιμοποιούμε ολόκληρη τη διαδρομή (full path) του αρχείου.
Εκτέλεση του uvspec.
Κατά την εκτέλεση του uvspec, το αρχείο χρειάζεται να διαβάσει μια σειρά από παραμετρικά αρχεία που βρίσκονται στον φάκελο της libRadtran. Η θέση των αρχείων αυτών μπορεί να δοθεί ως μία παράμετρος περιβάλλοντος (environmental variable). Αυτό γίνεται είτε ορίζοντάς την πριν την εκτέλεση, είτε βάζοντάς την στις ρυθμίσεις του shell του συστήματος.
export LIBRADTRAN_DATA_FILES='/location/of/libRadtran-2.0-32/data/'Κατά την εκτέλεση του uvspec είναι πρακτικό να χρησιμοποιούμε ένα κατάλληλα προετοιμασμένο αρχείο εισόδου (.inp) και να αποθηκεύουμε τα αποτελέσματα σε ένα αρχείο εξόδου (.out). Το πετυχαίνουμε αυτό κάνοντας χρήση της δυνατότητας ‘redirect’ της εισόδου (stdin “<”) και εξόδου (stdout “>”) του εκτελέσιμου.
uvspec < '/input/location/clear_atm.inp' > '/ouput/location/clear_atm.out'Παράλληλη εκτέλεση του ‘uvspec’ σε Bash shell.
Περισσότερα και πιο πρόσφατα αρχεία μπορούν να βρεθούν εδώ: github.com/thanasisn/IStillBreakStuff/tree/main/libradtran
Οι παρακάτω μέθοδοι έχουν δοκιμαστεί σε περιβάλλον GNU/Linux και Windows (Cygwin). Θεωρούμε ότι η εκτέλεση γίνεται σε έναν υπολογιστή του οποίου θέλουμε να αξιοποιήσουμε όλους ή μερικούς από τους πυρήνες του επεξεργαστή του. Με κάποιες τροποποιήσεις και σχεδίαση, οι λύσεις αυτές μπορούν να εφαρμοστούν ταυτόχρονα και σε περισσότερους υπολογιστές, δε θα αναφερθούμε ιδιαίτερα σε αυτό διότι υπάρχουν εξειδικευμένα εργαλεία για αυτή την δουλειά, όπως: GNU parallel (shell), PPSS (shell), HTCondor, dispy (python), snow (R) κ.λ.π.
Και στις δύο περιπτώσεις, πρέπει να υπάρχουν έτοιμα τα αρχεία εισόδου (.inp) της libRadtran. Ο τρόπος παραγωγής τους, δε θα μας απασχολήσει, καθώς μπορεί να γίνει με τα προτιμώμενα εργαλεία του χρήστη.
Παραλληλοποίηση με τη χρήση της εντολής xargs.
Σε αυτήν την περίπτωση χρησιμοποιούμε δύο Bash script, το πρώτο (xargs_uvspec_worker.sh) είναι υπεύθυνο για μία εκτέλεση του uvspec με τις κατάλληλες παραμέτρους (arguments). Το άλλο (xargs_parallel.sh), έχει την οργάνωση της παρτίδας εργασιών που θα εκτελεστούν από την εντολή xargs. Η εντολή xargs έχει την δυνατότητα να παρακολουθεί, μεταξύ άλλων, την χρήση των πυρήνων του επεξεργαστή, καθώς και να ελέγχει την εκτέλεση πολλαπλών εντολών.
Αρχείο xargs_uvspec_worker.sh
#!/bin/bash
## Worker to run one instance of uvspec, this is to be used by another script
## get arguments
OUTDIR="${1}"
ERRDIR="${2}"
total="${3}"
Tic="${4}"
INPUTF="${5}"
cntt="${6}"
## file to log this run
logfile="/path/to/a/log/file/JOB_$(date +"%F").log"
## set libradtran executable path
UVSPEC="/path/to/uvspec"
## check how many arguments
if [ $# -ne 6 ] ; then echo " 6 arguments needed" ; exit 1 ; fi
## input base file name
fname="$(basename $INPUTF)"
## out and error file names
OUTFIL="${OUTDIR}/${fname%.*}.out"
ERRFIL="${ERRDIR}/${fname%.*}.err"
## print some info while running
TOT=$(echo "scale=1; (($cntt*100/$total))" | bc)
ETA=$(($(($((total-cntt))*$(($(date +%s%N)-Tic))/60000000000))/cntt))
printf " %5s %5s/$total %5s%% ETA: %4s min\n" $((total-cntt)) $cntt $TOT $ETA
## keep a log of what happened
echo "$(date +"%F %T") $fname $cntt" >> "${logfile}"
#### HERE IS THE HEAVY LOAD ####
####TEST#### First try this to check what will run
echo "(( "${UVSPEC}" < "${INPUTF}") | gzip > "${OUTFIL}.gz") 2> ${ERRFIL}"
sleep $((RANDOM%5+2))
## Then use this to run the load
#(( "${UVSPEC}" < "${INPUTF}" ) | gzip > "${OUTFIL}.gz" ) 2> ${ERRFIL}
exit 0Αρχείο xargs_parallel.sh
#!/bin/bash
## Executioner of uvspec worker, this is used to run multiple script instances
## EDIT all paths to full paths
## this will run a uvspec
WORKER_sh="./xargs_uvspec_worker.sh"
## I/O folders
INPDIR="/path/to/files/for/INPUT/"
OUTDIR="/path/to/files/for/OUTPUT/"
ERRDIR="/path/to/files/for/error/"
## Cores to use
cores=8
####TEST#### DELETE THIS TEST VARIABLE
INPDIR="/home/.../LibRadTranM/clear_H2O_LAP/DATA"
## initial files count
total="$(find "${INPDIR}" -type f -iname "*.inp" | wc -l)"
## ask to continue
echo "" ; input=0
echo -n "Found $total input files continue (y/n)?: "
read -n 1 input ; echo
if [ "$input" == "y" -o "$input" == "Y" ] ; then
printf ""
else
echo "exit now.."; exit 2
fi
## set some variables
Tic="$(date +%s%N)" ## keep time
Tac="$(date +"%F %T")" ## keep time
cntt=0
#### THIS IS THE PARALLEL TRICK ####
## run all input files through the WORKER_sh
find "${INPDIR}" -type f -iname "*.inp" | sort | while read line;do
echo "$line" "$((++cntt))"
done | xargs -n 2 -P "$cores" "$WORKER_sh" "${OUTDIR}" \
"${ERRDIR}" "$total" "$Tic"
## you are done, print end report
T="$(($(date +%s%N)-Tic))"
S="$((T/1000000000))"
M="$((T%1000000000/1000000))"
echo ""
echo " ____UVSPEC_runs_finished____"
printf "DONE in: %02d %02d:%02d:%02d.%03d <\n" \
"$((S/86400))" "$((S/3600%24))" "$((S/60%60))" "$((S%60))" "${M}"
echo "From : $Tac"
echo "Until : $(date +"%F %T")"
exit 0Σημείωση: Τα προηγούμενα πιθανότατα θα τρέξουν στον mistral.
Προσοχή: Ακόμα και αν σταματήσει το κύριο script, οι workers θα συνεχίσουν να τρέχουν, οπότε πρέπει να ξέρετε πως μπορεί να σταματήσει ένα script που τρέχει στο background!
Παραλληλοποίηση με τη χρήση sub-shells στο background και της εντολής wait.
Εδώ κάνουμε χρήση μιας απλής τεχνικής ελέγχου του αριθμού των uvspec που εκτελούνται από τον υπολογιστή. Η λογική που εφαρμόζουμε είναι, να μετρούμε τις εκτελέσεις που έχουν γίνει και να περιμένουμε να τελειώσει κάποια από αυτές πριν ξεκινήσουμε την επόμενη.
Αρχείο execute_in_subshells.sh
#!/bin/bash
## Simple parallel execution in bash
## Test it before use. This can not be stopped with 'ctr+C'.
## All processes are sent to the background.
## libradtran executable
UVSPEC="/path/to/uvspec"
## folders
INPDIR="/path/to/files/for/INPUT/"
OUTDIR="/path/to/files/for/OUTPUT/"
ERRDIR="/path/to/files/for/error/"
LBRDAT="/path/to/libradtran/data/folder/data"
## file to log this run
logfile="/path/to/a/log/file/JOB_$(date +"%F_%T").log"
## parameters
cores=4 ## cores to use
prs=1 ## counter of concurrent processes
## ensure folders exist
mkdir -p "${OUTDIR}"
mkdir -p "${ERRDIR}"
## export libradtran data files path this may be redundant
export LIBRADTRAN_DATA_FILES="${LBRDAT}"
## count files
total="$(find "${INPDIR}" -type f -iname "*.inp" | wc -l)"
## ask to continue
echo "" ; input=0
echo -n "Found $total input files continue (y/n)?: "
read -n 1 input ; echo
if [ "$input" == "y" -o "$input" == "Y" ] ; then
printf ""
else
echo "exit now.."; exit 1
fi
cntt=0 ## count total runs
((cores--)) ## start from zero
Tic="$(date +%s%N)" ## keep time
Tac="$(date +"%F %T")" ## keep time
## list all input files
find "${INPDIR}" -type f -iname "*.inp" | while read line; do
((cntt++)) ## increase count
fname=$(basename "$line") ## input file-name
INPUT="$line" ## input file-name full path
OUTPUT="$OUTDIR/${fname%.*}.OUT" ## output file-name full path
ERROR="$ERRDIR/${fname%.*}.err" ## error file-name full path
## print some info
TOT=$(echo "scale=1; (($cntt*100/$total))" | bc)
ETA=$(($(($((total-cntt))*$(($(date +%s%N)-Tic))/60000000000))/cntt))
printf " %5s %5s/$total %5s%% prs: %2s ETA: %s min\n" \
$((total-cntt)) $cntt $TOT $prs $ETA
## keep a log of what happened
echo "$(date +"%F %T") $fname $cntt" >> "${logfile}"
#### uncomment to choose output with gzip compression
# ( ( ( "${UVSPEC}" < "${INPUT}" ) | gzip > "${OUTPUT}.gz" ) 2> $ERROR ) &
#### uncomment for output without compression
# ( ( ( "${UVSPEC}" < "${INPUT}" ) > "${OUTPUT}" ) 2> $ERROR ) &
#### uncomment for output without compression and
#### export libradtran data path this may be redundant
# ( ( ( export LIBRADTRAN_DATA_FILES="${LBRDAT}"
# "${UVSPEC}" < "${INPUT}" ) > "${OUTPUT}" ) 2> $ERROR ) &
## Throttle execution. This keeps script from running everything at once!
if (( ++prs > cores )); then
wait
((prs--))
fi
done
## wait for the last of the runs after the loop ends
for i in $(seq 1 $cores); do; wait; done
## print end report
T="$(($(date +%s%N)-Tic))"
S="$((T/1000000000))"
M="$((T%1000000000/1000000))"
echo ""
echo " ____UVSPEC_runs_finished____"
printf "DONE in: %02d %02d:%02d:%02d.%03d <\n" \
"$((S/86400))" "$((S/3600%24))" "$((S/60%60))" "$((S%60))" "${M}"
echo "From : $Tac"
echo "Until : $(date +"%F %T")"
exit 0Σημείωση: Αυτό πιθανότατα δε θα τρέξει στον mistral λόγω διαφορών στις εκδόσεις της wait. Δουλεύει σε διανομές Debian.
Προσοχή: Για να το σταματήσετε πρέπει να τερματίσετε όλες τις διεργασίες που γίνονται στο background και όχι μόνο αυτό το script (ctrl+C). Γι’ αυτό πρέπει να ξέρετε πώς τερματίζονται εργασίες στον background.
Παραλληλοποίηση με τη χρήση του GNU parallel.
Με το parallel (GNU parallel) μπορούμε να χρησιμοποιήσουμε πολλαπλούς πυρήνες ενός υπολογιστή, αλλά και περισσότερους από έναν υπολογιστή (cluster). Το parallel είναι στην ουσία ένας διαχειριστής εκτελέσεων (job scheduler). Τα επόμενα παραδείγματα είναι ίσως η πιο απλοποιημένη παραμετροποίηση που μπορούμε να κάνουμε. Για πιο σύνθετες περιπτώσεις δείτε τα εγχειρίδια των προγραμμάτων.
Παραμετροποίηση cluster.
Για να έχει πρόσβαση το parallel στους υπόλοιπους υπολογιστές, χρειάζεται πρώτα να ρυθμίσουμε το ssh ώστε να μπορεί να βρει του υπόλοιπους υπολογιστές στο δίκτυο.
Configure network.
Οι υπολογιστές μπορεί να βρίσκονται στο τοπικό μας δίκτυο ή να έχουν σταθερή διεύθυνση IP, οπότε χρειάζεται ελάχιστη ρύθμιση. Είτε, να έχουμε ρυθμισμένο κάποιο VPN ώστε να μη χρειάζεται να μας απασχολεί η περίπτωση της μη σταθερής διεύθυνση IP ή της μετακίνησης του υπολογιστή σε άλλο δίκτυο (π.χ. laptop).
Configure ssh.
Πρέπει να έχουμε πρόσβαση σε κάθε υπολογιστή μέσω ssh και τη χρήση κλειδιού χωρίς την ανάγκη εισαγωγής κωδικού. Η διαδικασία ρύθμισης είναι εύκολο να βρεθεί στο internet ψάχνοντας π.χ. “passwordless authentication ssh keys”. Μετά από αυτό πρέπει να μπορείτε να έχετε πρόσβαση σε κάθε έναν από τους υπολογιστές εκτελώντας την εντολή π.χ. ssh user@155.207.10.10 -i .ssh/libradtran_cl.pri χωρίς να εισάγεται κωδικό. Όπου “155.207.10.10” είναι η θέση του υπολογιστή στο δίκτυο και “libradtran_cl.pri” το αρχείο του προσωπικού κλειδιού πρόσβασης (private key).
Για να οργανώσουμε το cluster χρειάζεται να συμπληρώσουμε ένα παραμετρικό αρχείο του ssh όπου θα αναφέρονται όλοι οι υπολογιστές που θέλουμε να χρησιμοποιήσουμε (default file .ssh/config). Αν όλα γίνουν σωστά θα μπορούμε να συνδεόμαστε μέσω ssh δίνοντας μόνο το όνομα του υπολογιστή π.χ. ssh machine_1. Αυτό, επίσης βοηθάει πολύ, στη γενικότερη αντιμετώπιση των προβλημάτων που μπορεί να προκύψουν, αλλά και στην παραμετροποίηση του κάθε υπολογιστή για την δουλειά που σκοπεύουμε να τρέξουμε.
Παραμετροποίηση του parallel
Αφού ο κεντρικός υπολογιστής έχει πρόσβαση σε όλους του άλλους μέσω ssh, αρκεί ένα απλό παραμετρικό αρχείο για το parallel, με του υπολογιστές που θέλουμε να χρησιμοποιήσουμε στο cluster.
Στο παρακάτω αρχείο, δηλώνουμε ότι το cluster θα έχει τέσσερις υπολογιστές. Με το σύμβολο : είναι ο κεντρικός υπολογιστής, όπου οι πυρήνες του αναγνωρίζονται αυτόματα. Ο αριθμός πριν το όνομα του υπολογιστή δηλώνει τους πυρήνες του επεξεργαστή που θα είναι διαθέσιμοι από τον κάθε υπολογιστή.
Χρήση parallel
Ένα παράδειγμα εκτέλεσης του parallel. Η χρήση των παραμέτρων αναλύεται στο manual της εντολής.
Εδώ χρησιμοποιούμε έναν πυρήνα από κάθε υπολογιστή του cluster, όπου θα τρέξουμε τις εντολές που βρίσκονται στο αρχείο jobs.list. Όπου το αρχείο optimise_worker_v1.R παίρνει δύο παραμέτρους που τις χρησιμοποιεί για να τρέξει ένα εύρος παραμέτρων από μία άλλη λίστα εργασιών. Έτσι έχουμε χωρίσει την συνολική εργασία σε εκτελέσεις 100 μικρότερων εργασιών που αναθέτουμε σε κάθε υπολογιστή του cluster.
parallel \
--jobs 1 \
--progress \
--eta \
--results /home/athan/Aerosols_O3/DATA/par.out \
--joblog /home/athan/Aerosols_O3/Libradtran_modeling/par.resume.file \
--sshloginfile ~/.parallel/hosts \
-a /home/athan/Aerosols_O3/Libradtran_modeling/jobs.listΑρχείο jobs.list.
/home/athan/Aerosols_O3/Libradtran_modeling/optimise_worker_v1.R 1 100
/home/athan/Aerosols_O3/Libradtran_modeling/optimise_worker_v1.R 101 200
/home/athan/Aerosols_O3/Libradtran_modeling/optimise_worker_v1.R 201 300
/home/athan/Aerosols_O3/Libradtran_modeling/optimise_worker_v1.R 301 400
/home/athan/Aerosols_O3/Libradtran_modeling/optimise_worker_v1.R 401 500
/home/athan/Aerosols_O3/Libradtran_modeling/optimise_worker_v1.R 501 600
/home/athan/Aerosols_O3/Libradtran_modeling/optimise_worker_v1.R 601 700
/home/athan/Aerosols_O3/Libradtran_modeling/optimise_worker_v1.R 701 800
/home/athan/Aerosols_O3/Libradtran_modeling/optimise_worker_v1.R 801 900
/home/athan/Aerosols_O3/Libradtran_modeling/optimise_worker_v1.R 901 1000Συγχρονισμός/μεταφορά αρχείων.
Ανάλογα με τη λογική της εφαρμογής της παραλληλοποίησης, είναι λιγότερο η περισσότερο απαραίτητο να μετακινούνται αρχεία μεταξύ των υπολογιστών. Αυτά τα αρχεία μπορεί να είναι αρχεία προς επεξεργασία, εκτελέσιμα ή αρχεία αποτελεσμάτων. Το parallel έχει κάποιες τέτοιες δυνατότητες, αλλά είναι ίσως πιο εύκολο να χρησιμοποιηθεί κάποια πιο εξειδικευμένη εφαρμογή. Δε θα αναλύσουμε την εφαρμογή τους αλλά θα δώσουμε κάποιες ιδέες.
unison: Αμφίδρομα συγχρονίζει αρχεία μεταξύ υπολογιστών (όσα χρειάζονται), πολλές δυνατότητες παραμετροποίησης.
rsync: Συγχρονίζει αρχεία μεταξύ υπολογιστών (όσα χρειάζονται), βελτιστοποιημένο στη μεταφορά αρχείων.
owncloud: Cloud server απόθεσης αρχείων, μπορεί να εγκατασταθεί σε οποιονδήποτε υπολογιστή και οι υπόλοιποι να συγχρονίζονται από αυτόν.
nfs: Δίσκος δικτύου, στον οποίο συνδέονται όλοι οι υπολογιστές και τον χρησιμοποιούν ως τοπικό.
Προφανώς, υπάρχουν και άλλοι τρόποι, και όποια επιλογή εξαρτάται από την διάρθρωση του δικτύου, τους διαθέσιμους πόρους και τον επιθυμητό στόχο.
Εκτέλεση της libRadtran σε HPC cluster.
Εισαγωγή
Εάν έχουμε την δυνατότητα, να χρησιμοποιήσουμε το HPC grid του πανεπιστημίου, μπορούμε να εκτελέσουμε πολύ μεγάλο όγκο προσομοιώσεων της ακτινοβολίας στην ατμόσφαιρα με την libRadtran, σε μικρό χρονικό διάστημα. Για τον σκοπό αυτό φτιάξαμε μερικά εργαλεία, τα οποία βοηθούν στη χρήση του συστήματος Portable Batch System (PBS/Torque) που ελέγχει την εκτέλεση και τον καταμερισμό των εργασιών στο grid.
Γενικά, θα αναφερόμαστε σε μία εκτέλεση του uvspec (εκτελέσιμο αρχείο της libRadtran) ως μία ανεξάρτητη ‘εργασία’ (job), αφού αυτή εξαρτάται μόνο από μία προκαθορισμένη ομάδα παραμέτρων. Αυτές οι ομάδες παραμέτρων αποτελούν μία λίστα πολλαπλών εργασιών τις οποίες αναθέτουμε στο PBS να εκτελέσει. Θα αναφερόμαστε σε αυτές ως παρτίδα εργασιών (batch job).
Μπορούμε να χωρίσουμε την όλη διαδικασία σε τρεις φάσεις.
- Δημιουργία της λίστα παραμέτρων προς εκτέλεση.
- Ανάθεση της εκτέλεσης στο PBS.
- Επεξεργασία/διαχείριση των αποτελεσμάτων.
Ανάλογα με τις ανάγκες, οι φάσεις αυτές επαναλαμβάνονται κυκλικά μέχρι να έχουμε τα επιθυμητά δεδομένα. Γι’ αυτό, είναι καλό να λάβουμε κάποια μέτρα κατά την προετοιμασία της ακολουθίας των φάσεων (workflow) ώστε να αποφύγουμε προβλήματα και να διευκολύνουμε την παραγωγή και διαχείριση των αποτελεσμάτων. Δε θα αναφερθούμε ιδιαίτερα σε αυτό, αλλά έχουμε φροντίσει ώστε να υπάρχει επαρκές logging και παρακολούθηση σε όλα τα στάδια της διαδικασίας ώστε να μπορούν να εντοπιστούν εύκολα τα προβλήματα και να διορθωθούν.
Ανάθεση της εκτέλεσης στο PBS grid.
Εκτέλεση της εντολής uvspec (libRadtran) ως μία εργασία (job).
Χρησιμοποιούμε ένα script (LBT_PBS.sh) σε Bash το οποίο αναλαμβάνει να καλέσει το εκτελέσιμο uvspec με τις παραμέτρους που του δίνει το σύστημα PBS. Αυτό το αρχείο θα εκτελεστεί σε κάθε πυρήνα, από τους διαθέσιμους και είναι υπεύθυνο για την ολοκλήρωση μίας προσομοίωσης ακτινοβολίας του μοντέλου.
Οι παράμετροι (arguments) που δέχεται αυτό το script είναι τέσσερις.
- Libradtran file path: ο φάκελος όπου βρίσκονται τα αρχεία της libRadtran.
- Libradtran output folder: ο φάκελος όπου θα αποθηκευτούν τα παραγόμενα αρχεία.
- unique id: μοναδικός δείκτης για να χρησιμοποιηθεί στο όνομα των αρχείων κάθε εργασίας.
- serialized string with uvspec options: μία λέξη με όλες τις παραμέτρους που χρειάζεται να ορίσουμε σε μία εκτέλεση του
uvspec.
Ο τρόπος που έχουμε επιλέξει για την αποστολή των παραμέτρων στο uvspec είναι η γραμμικοποίησή τους (serialization). Αυτό επιτρέπει την χρήση οποιοδήποτε κατάλληλων παραμέτρων της libRadtran χωρίς καμία τροποποίηση αυτού του αρχείου. Αυτό το πετυχαίνουμε, έχοντας κωδικοποιήσει την ερμηνεία του συμβόλου @ ως νέα γραμμή και του @@ ως τον χαρακτήρα του κενού. Με αυτόν τον τρόπο κάθε αρχείο εισόδου (.inp) μπορεί να σταλεί ως μία λέξη. Για παράδειγμα, η λέξη:
atmosphere_file@@aattmmoo=afglms.dat@source@@solar@@ssoollaa=kurudz_1.0nm.
dat@mol_modify@@O3@@290@@DU@albedo@@0.05@sza@@26@altitude@@0.062694168@rte
_solver@@sdisort@number_of_streams@@6@wavelength@@250@@5025@pseudospherica
l@quiet@θα μετατραπεί στο αρχείο εισόδου (.inp):
atmosphere_file /mnt/.../libRadtran-2.0.1/data/atmmod/afglms.dat
source solar /mnt/.../libRadtran-2.0.1/data/solar_flux/kurudz_1.0nm.dat
mol_modify O3 290 DU
albedo 0.05
sza 26
altitude 0.062694168
rte_solver sdisort
number_of_streams 6
wavelength 250 5025
pseudospherical
quiet
Αρχείο LBT_PBS.sh.
Στο πρώτο κομμάτι του script ελέγχουμε αν όλες οι παράμετροι έχουν δοθεί κανονικά από το PBS και προετοιμάζουμε τα ονόματα των αρχείων με την πλήρη τους διαδρομή στο δίσκο.
#!/bin/bash
libpath=$1 ## Libradtran file path .../libRadtran-2.0/
workdir=$2 ## Libradtran output folder for this model family
jobid=$3 ## unique id for this run
options=$4 ## serialized string with uvspec options
## expand file paths
UVSPEC="${libpath}/bin/uvspec"
DATA="${libpath}/data"
WPTEMP="${workdir}/clearwaterLAP"
## have libRadtran path?
if [ -z "$libpath" ]; then
echo "Empty variable 'libpath'"
exit 2
fi
## check executable location
if [ ! -f "$UVSPEC" ]; then
echo "Can not find uvspec" ; exit 3
fi
## check data folder location
if [ ! -d "$DATA" ]; then
echo "Can not find data folder $DATA" ; exit 4
fi
## have working dir path?
if [ -z "$workdir" ]; then
echo "Empty variable 'workdir'" ; exit 5
fi
## check working folder location
if [ ! -d "$workdir" ]; then
echo "Can not find working folder $workdir" ; exit 6
fi
## have jobid?
if [ -z "$jobid" ]; then
echo "Empty variable 'jobid'" ; exit 7
fi
## have libratran options?
if [ -z "$options" ]; then
echo "Empty variable 'options'" ; exit 8
fi
## create working folder
mkdir -p "${WPTEMP}"
## files for this job
INPUT="${WPTEMP}/LBT_${jobid}.inp"
OUPUT="${WPTEMP}/LBT_${jobid}.out"
ERPUT="${WPTEMP}/LBT_${jobid}.err"Στη συνέχεια μορφοποιούμε τις παραμέτρους της libRadtran σε ένα κατάλληλο αρχείο “.inp”.
## create input file
( echo $options | sed 's/@@/ /g' | sed 's/@/\n/g' | while read line ;do
echo $line | sed "s@aattmmoo=@"$DATA/atmmod/"@g" \
| sed "s@ssoollaa=@"$DATA/solar_flux/"@g"
done ) > $INPUTΕκτελούμε το uvspec παραπέμποντας τα κατάλληλα αρχεία (εισόδου, εξόδου και σφαλμάτων) και όταν τελειώσει η εκτέλεσή του συμπιέζουμε το αρχείο εξόδου. Η συμπίεση βοηθάει στο να μειωθεί το μέγεθος των αρχείων αποτελεσμάτων αλλά και στη μεταφορά τους από τον υπολογιστή που έκανε την εκτέλεση, στον υπολογιστή ελέγχου.
#### ready to run uvspec
tic=$(date +"%s")
loa="$(uptime | grep -o "load .*")"
export LIBRADTRAN_DATA_FILES=${DATA}
( $UVSPEC < $INPUT > $OUPUT ) >& $ERPUT
wait; wait
gzip -f $OUPUT
tac=$(date +"%s")Αποθηκεύουμε κάποιες επιπρόσθετες πληροφορίες σχετικά με τον χρόνο εκτέλεσης και το σύστημα στο οποίο έγινε, ώστε να τις χρησιμοποιήσουμε σε περίπτωση σφαλμάτων.
## helpful info for this run and uvspec error collector
( echo $loa
uptime | grep -o "load .*"
echo "hostname=$(hostname)"
date +"%F %T"
echo $tic
echo $tac ) >> $ERPUT
exit 0Τροφοδοσία παρτίδας εργασιών στο grid.
Για να αναθέσουμε μία παρτίδα εργασιών (batch job), χρησιμοποιούμε ένα παραμετρικό αρχείο (submit.pbs) το οποίο περιέχει και ορίζει τις παραμέτρους εκτέλεση της παρτίδας. Εδώ, καθορίζονται οι παράμετροι που ελέγχουν τον τρόπο εκτελεσης της λίστα εργασιών. Για παράδειγμα, το πώς θα γίνει η αναφορά κατά την εκτέλεση, η διαχείριση των λαθών (error handling) κλπ.
Το αρχείο αυτό έχει ταυτόχρονα την λειτουργία του παραμετρικού αρχείου για το PBS. Αλλά, και την λειτουργία Bash script που το εκτελεί το PBS προκειμένου να χρησιμοποιήσει το περιεχόμενό/αποτέλεσμα των εντολών του.
Το δείγμα αρχείου που παραθέτουμε μπορεί να χρησιμοποιηθεί, είτε για την μερική εκτέλεση/δοκιμή μιας παρτίδας εργασιών, ή για την πλήρη εκτέλεση μιας παρτίδας.
Αρχείο submit.pbs.
Οι σειρές που ξεκινούν με ‘#PBS’ αφορούν παραμέτρους (arguments) που χρησιμοποιούνται από την εντολή qsub του PBS. Περισσότερες λεπτομέρειες στο manual της εντολής (man qsub).
#!/bin/bash
#PBS -N clearatm
#PBS -j oe
#PBS -q see
#PBS -M at...ys@gmail.com
#PBS -m abe
#PBS -l nodes=1:ppn=1
#PBS -o condorlog/run.log
#PBS -t 1-10Οι μεταβλητές που ξεκινούν με PBS_* παίρνουν τιμή από το σύστημα PBS κατά την εκτέλεση των ανεξάρτητων εργασιών (jobs). Εδώ, ως εργασία νοείται η εκτέλεση του αρχείου LBT_PBS.sh με τις κατάλληλες παραμέτρους και φυσικά αυτό είναι που θα καλέσει τελικά το uvspec. Όλα τα υπόλοιπα καθορίζουν την ανάσυρση των παραμέτρων από την λίστα εργασιών και η καταγραφή της προόδου της παρτίδας σε αρχείο.
jobhomedir="/mnt/.../LibRadTranM/clear_water_pressure_ozone_LAP_meas/"
joblogfile="${jobhomedir}/condorlog/clearwatermeas_$(date +"%F").log"
echo $PBS_O_WORKDIR >> "${joblogfile}"
cd "$jobhomedir"
## parse arguments from file
args=`sed -n "${PBS_ARRAYID} p" jobs_args.list`
arglist=($args)
arg1="/mnt/lapmg_a/.../libRadtran-2.0.1/"
arg2="/mnt/lapmg_a/.../LibRadTranM/clear_water_pressure_ozone_LAP_meas/"
arg3=${arglist[0]}
arg4=${arglist[1]}
( echo $(hostname) $PBS_O_HOST $PBS_SERVER $PBS_O_QUEUE
echo ${PBS_O_WORKDIR}
echo "JOBID =" ${PBS_ARRAYID}
echo "arg12" ${arg1} ${arg2}
echo "arg3="${arg3}
echo "arg4="${arg4}
echo "-~-~-~-~-~-~-~-~-~-~-~-~-~-~-~-~-~-~-~-~-~-~-~-~-~-~-~"
) >> "${joblogfile}"
## this is a job run
./LBT_PBS.sh $arg1 $arg2 $arg3 $arg4Θεωρούμε ότι ο αναγνώστης, είτε είναι εξοικειωμένος με τη χρήση του Bash shell, είτε ότι μπορεί να αναζητήσει πληροφορίες για την χρήση και την ερμηνεία των παραπάνω εντολών στο διαδίκτυο.
Εκτέλεση μεγάλης παρτίδας
Προκειμένου να οργανώσουμε καλύτερα την κατάθεση (submission) κάποιας μεγάλης παρτίδας (1000+ jobs), και κατόπιν επικοινωνίας με τους διαχειριστές του grid. Για την καλύτερη εξυπηρέτηση των χρηστών του grid, γράψαμε ένα Bash script ώστε να κάνουμε την κατάθεση σε μικρότερα τμήματα και με κάποια χρονική απόσταση.
Αρχείο multisub.sh
Οι μεταβλητές ‘step’ και ‘watt’ καθορίζουν αντίστοιχα το μέγεθος των πακέτων εργασιών και τον χρόνου μεταξύ κάθε τμήματος. Οι τιμές τους σχετίζονται με τον χρόνο εκτέλεσης της libRadtran και τους διαθέσιμους πυρήνες. Οι τιμές εδώ, είναι ενδεικτικές για “σχετικά βαριές” εκτελέσεις της libRadtran. Σημειώνουμε ότι εδώ ο χρόνος αναμονής μεταξύ των πακέτων αυξάνεται γραμμικά.
#!/bin/bash
subfile="clearatm_submit.pbs"
total=4900
step=150
watt=40
## get total lines
total="$(cat jobs_args.list | wc -l)"
echo
echo $subfile
echo Total: $total
echo step : $step
echo wait : $watt
echo "" ; input=0
echo -n "Submit (y/n)?: "
read input
if [ "$input" == "y" -o "$input" == "Y" ] ; then
echo " Bombs Away...."
echo
else
echo
echo "ABORT! ABORT! ABORT! ABORT!"
echo
exit 0
fi
echo
for ii in $(seq 0 $((total / step - 1))); do
date -d"+$((watt*ii+1)) seconds" +"next at: %F %T "; echo
sleep $((watt*ii+1))
echo "running: qsub -t $((step*ii+1))-$((step*ii+step)) ${subfile}"
qsub -t $((step*ii+1))-$((step*ii+step)) "${subfile}"
done
if [ $((total % step )) -gt 0 ];then
date -d"+$((watt*ii+1)) seconds" +"next at: %F %T "; echo
sleep $((watt*ii+1))
echo "run: qsub -t $((step*ii+step+1))-$((step*ii+step+total%step)) ${subfile}"
qsub -t $((step*ii+step+1))-$((step*ii+step+total%step)) "${subfile}"
fi
echo
echo " * * * submitting done * * *"
echo
exit 0Κοινές εντολές για την διαχείριση των παρτίδων εργασιών.
Εκτέλεση παρτίδας όπως καθορίζεται από τις παραμέτρους του αρχείου ‘submit.pbs’. Η γραμμή ‘#PBS -t 1-10’ στο αρχείο λέει στο PBS να εκτελέσει τις εργασίες 1 έως 10. Αυτός είναι ένας τρόπος για να δοκιμάζονται μεγαλύτερες παρτίδες εργασιών ως προς την ορθότητα των ρυθμίσεών τους, πριν αφεθούν να τρέξουν στο σύνολό τους.
qsub submit.pbsΕνας τρόπος να μπουν 100 εργασίες στη σειρά αναμονής προς εκτέλεση (queue).
qsub -t 301-400 submit.pbsΛίστα με τις εργασίες στην ουρά (queue).
qstat -tΛίστα με τις εργασίες που εκτελούνται.
qstat -eΛίστα με τους υπολογιστές (nodes) όπου εκτελούνται οι εργασίες.
qstat -n1Σταματάει όλες τις εργασίες της παρτίδας 4168020.
qdel 4168020Σταμάτημα επιλεγμένων εργασιών της παρτίδας 4168020.
qdel 4168020[]Συνεχείς τρόποι παρακολούθησης των εργασιών σε εκτέλεση/ουρά.
watch -ν10 ' qstat -t | grep " R " | wc -l ; \
qstat -t | grep " Q " | wc -l ; \
qstat -t '
watch ' qstat -t | grep " R " | wc -l ; \
qstat -t | grep " Q " | wc -l ; \
qstat -t | grep " R " 'Διαθέσιμοι πόροι χωρίς τους εκτός λειτουργίας κόμβους.
pbsnodes -cΛίστα όλων των κόμβων.
pbsnodes -lΠερισότερες πληροφορίες για τις παραπάνω εντολές μπορούν να βρεθούν στις αντίστοιχες
σελίδες manual (man pbsnodes, man qstat, man qsub) ή στο Παράρτημα (pbsnodes: ??,
qstat: ??,
qsub: ??).
Επεξεργασία των αποτελεσμάτων.
Ο υπολογιστής από τον οποίο κάνουμε την ανάθεση των εργασιών έχει εγκατεστημένη την γλώσσα προγραμματισμού ‘R’. Θα την χρησιμοποιήσουμε για να συγκεντρώσουμε τα αποτελέσματα που μας ενδιαφέρουν.
Θα διαβάσουμε τα πρωτογενή δεδομένα από τα αρχεία που παράχθηκαν κατά την εκτέλεση της libRadtran και θα τα αποθηκεύσουμε σε ένα αρχείο δεδομένων της ‘R’. Με αυτόν τον τρόπο, αποφεύγουμε την μεταφορά μεγάλων ποσοτήτων δεδομένων, στις περιπτώσεις που μας ενδιαφέρουν μόνο κάποιες συγκεντρωτικές τιμές.
Παρακάτω παρατίθεται ένα παράδειγμα για το πώς μπορεί να επιτευχθεί αυτό. Δεν κρίνουμε σκόπιμο να αναλύσουμε τη λειτουργία του script διότι η διαδικασία αυτή μπορεί να γίνει με διαφορετικού τρόπους και χρησιμοποιώντας πολλές άλλες γλώσσες προγραμματισμού, ανάλογα με τις ανάγκες και τις δεξιότητες του χρήστη.
Αρχείο parse_data.R.
#!/usr/bin/env Rscript
#### Clear environment
#### ----------------------------------------------
closeAllConnections()
rm(list = (ls()[ls() != ""]))
Sys.setenv(TZ = "UTC")
tic <- Sys.time()
## folder for this model family
## setwd('/mnt/lapmg_a/.../LibRadTranM/lear_H2O_LAP')
## folder paths
jobsfolder <- "clearwaterLAPmeas/"
datafolder <- "DATA/"
archfolder <- "DATA/ARCHIVED/"
if (file.exists(datafolder) &
file.exists(archfolder) &
file.exists(jobsfolder)) {
cat("\nFolders exists\n")
} else {
stop("Can not see all Folders")
}
## read libraries
source("../R_common/trapezUVSPEC.R")
## data output
datafile <- "../clear_H2O_PR_O3_meas_LAP.Rds"
## function to read inp files
input_parms <- function(inputfile) {
if (!file.exists(inputfile))
stop(paste("Missinig file: ", inputfile))
# read input file
fcon <- file(inputfile)
lines <- readLines(fcon)
close(fcon)
# 1
atmosphere_file <- basename(
unlist(
strsplit(
grep("atmosphere_file", lines, value = TRUE),
" +"
)
)[2]
)
atmosphere_file <- unlist(strsplit(atmosphere_file, ".dat", fixed = T))[1]
# 2
source_solar <- basename(
unlist(
strsplit(
grep("source solar", lines, value = TRUE),
" +"
)
)[3]
)
source_solar <- unlist(strsplit(source_solar, ".dat", fixed = T))[1]
# 3
mol_modify_O3 <- as.numeric(
unlist(
strsplit(
grep("mol_modify O3", lines, value = TRUE),
" +"
)
)[3]
)
# 4
albedo <- as.numeric(
unlist(
strsplit(
grep("albedo", lines, value = TRUE),
" +"
)
)[2]
)
# 5
sza <- as.numeric(
unlist(
strsplit(
grep("sza", lines, value = TRUE),
" +"
)
)[2]
)
# 6
altitude <- as.numeric(
unlist(
strsplit(
grep("altitude", lines, value = TRUE),
" +"
)
)[2]
)
# 7
rte_solver <- unlist(
strsplit(
grep("rte_solver", lines, value = TRUE),
" +"
)
)[2]
# 8
number_of_streams <- as.numeric(
unlist(
strsplit(
grep("number_of_streams", lines, value = TRUE),
" +"
)
)[2]
)
# 9
wavelength <- as.numeric(
unlist(
strsplit(
grep("wavelength", lines, value = TRUE),
" +"
)
)[2:3]
)
wvlngth_min <- wavelength[1]
wvlngth_max <- wavelength[2]
# 10
pseudospherical <- any(grepl("^ *pseudospherical", lines))
# 11
mol_modify_H2O <- as.numeric(
unlist(
strsplit(
grep(
"mol_modify H2O +[.0-9]+ +MM", lines,
value = TRUE
),
" +"
)
)[3]
)
# 12
pressure <- as.numeric(
unlist(
strsplit(
grep("pressure", lines, value = TRUE),
" +"
)
)[2]
)
aninput <- data.frame(
atmosphere_file = atmosphere_file, source_solar = source_solar,
mol_modify_O3 = mol_modify_O3, mol_modify_H2O = mol_modify_H2O,
albedo = albedo, sza = sza, altitude = altitude,
pressure = pressure, rte_solver = rte_solver, number_of_streams = number_of_streams,
wvlngth_min = wvlngth_min, wvlngth_max = wvlngth_max
)
return(aninput)
}
## function to get integral of .out collumns
output_read_trapz <- function(outputfile) {
if (!file.exists(outputfile))
stop(paste("Missinig file: ", outputfile))
## fmt: lambda edir edn eup uavgdir uavgdn uavgup
tempdata <- read.table(outputfile)
get <- trapezUVSPEC(tempdata)
data.frame(
edir = get[1], edn = get[2], eup = get[3], eglo = get[1] +
get[2]
)
}
## function to read .err files
error_param <- function(errfile) {
if (!file.exists(errfile))
stop(paste("Missinig file: ", errfile))
# read input file
fcon <- file(errfile)
lines <- readLines(fcon)
close(fcon)
## start end
minD <- min(as.numeric(grep("^[0-9]+$", lines, value = TRUE)))
maxD <- max(as.numeric(grep("^[0-9]+$", lines, value = TRUE)))
hosts <- grep("hostname=.*", lines, value = TRUE)
hosts <- unlist(strsplit(hosts, "="))[2]
if (hosts != "") {
host <- hosts
} else {
host <- "unknown"
}
return(
data.frame(hostname = host, ticTime = minD, tacTime = maxD)
)
}
#### Parsing starts here ##### read saved data or fail
saved_data <- readRDS(datafile)
# saved_data <- data.frame() # if there is no previous
# file read list of inp files inpfiles =
# list.files(path = datafolder,
inpfiles <- list.files(
path = jobsfolder, pattern = "LBT_.*.inp", full.names = TRUE,
recursive = FALSE
)
if (length(inpfiles) <
1) stop("No input files found")
## check if all files matching
pause <- FALSE
for (ii in inpfiles) {
outputfile <- paste0(
unlist(strsplit(ii, split = ".inp"))[1],
".out.gz"
)
errfile <- paste0(
unlist(strsplit(ii, split = ".inp"))[1],
".err"
)
## check output files
if (!file.exists(outputfile)) {
cat(
paste("Missing file", outputfile),
sep = "\n"
)
pause <- TRUE
}
## check error files
if (!file.exists(errfile)) {
cat(
paste("Missing file", errfile),
sep = "\n"
)
pause <- TRUE
}
}
outfiles <- list.files(
path = jobsfolder, pattern = "LBT_.*.out.gz", full.names = TRUE,
recursive = FALSE
)
for (ii in outfiles) {
inpurfile <- paste0(
unlist(strsplit(ii, split = ".out.gz"))[1],
".inp"
)
## check input files
if (!file.exists(inpurfile)) {
cat(
paste("Missing file", inpurfile),
sep = "\n"
)
pause <- TRUE
}
}
if (pause) {
stop("Pause to manual clean files")
}
cat(
paste(
length(inpfiles),
" files to read\n"
)
)
## read data to a data frame
gather <- data.frame()
ccc <- 0
for (ii in inpfiles) {
ccc <- ccc + 1
cat(
paste(
ccc, "/", length(inpfiles),
" processed\n"
)
)
outputfile <- paste0(
unlist(strsplit(ii, split = ".inp"))[1],
".out.gz"
)
errfile <- paste0(
unlist(strsplit(ii, split = ".inp"))[1],
".err"
)
record <- cbind(
input_parms(ii),
output_read_trapz(outputfile),
error_param(errfile)
)
gather <- rbind(gather, record)
}
colall <- c(
"atmosphere_file", "source_solar", "mol_modify_O3",
"albedo", "sza", "altitude", "rte_solver", "number_of_streams",
"wvlngth_min", "wvlngth_max", "edir", "eglo", "edn",
"eup", "mol_modify_H2O", "pressure"
)
colinp <- c(
"atmosphere_file", "source_solar", "mol_modify_O3",
"albedo", "sza", "altitude", "rte_solver", "number_of_streams",
"wvlngth_min", "wvlngth_max", "mol_modify_H2O", "pressure"
)
## combine old and new data
combined_data <- rbind(saved_data, gather)
## keep unique input combination
uniqueinqx <- !duplicated(combined_data[, colinp])
combined_data <- combined_data[uniqueinqx, ]
## check different results for same input
if (any(duplicated(combined_data[, colall]))) {
stop("Posible different results for the same input")
}
# ## this will remove stored data!! saved_data =
# saved_data[ saved_data$sza<50, ] saveRDS(
# saved_data, file = datafile, compress = 'xz') save
# this set of data as local repository combined_data
# <- data.frame() ## used to reset stored data
saveRDS(combined_data, file = datafile, compress = "xz")
cat(" now you can move files to ARCHIVED ")
# print(apply(combined_data[,c(1:10,17)], 2, unique))
str(combined_data)
summary(combined_data)Δημιουργία λίστα παραμέτρων προς εκτέλεση.
Η λίστα παραμέτρων μίας παρτίδας εργασιών περιγράφει όλες τις συνθήκες με τις οποίες επιθυμούμε να γίνει η προσομοίωση της ακτινοβολίας στην ατμόσφαιρα.
Επιλέξαμε να δημιουργούμε αυτή τη λίστα σε δύο στάδια. Στην αρχή, καθορίζονται οι συνθήκες για τις οποίες επιθυμούμε να τρέξουμε την libRadtran και από αυτές κατασκευάζουμε όλα τα ενδεχόμενα. Στη συνέχεια, η λίστα των επιθυμητών ενδεχομένων συγκρίνεται με τα υπάρχοντα αποτελέσματα, ώστε να αποφευχθεί ο επαναυπολογισμός ήδη έτοιμων δεδομένων. Το αποτέλεσμα είναι μία νέα παρτίδα εργασιών που μπορεί να κατατεθεί στο grid για επεξεργασία.
Με αυτόν τον τρόπο, μπορούμε να ξεκινήσουμε με αρκετά αραιές επιθυμητές συνθήκες, και στη συνέχεια να αυξήσουμε την πυκνότητά τους ανάλογα με τις παραμέτρους για τις οποίες ενδιαφερόμαστε. Μοιράζοντας τον φόρτο εργασίας σε μικρότερα κομμάτια, έχοντας τη δυνατότητα να εκτιμήσουμε προοδευτικά τα αποτελέσματα που παίρνουμε από το μοντέλο και ελέγχοντας για την ύπαρξη προηγούμενων αποτελεσμάτων, μπορούμε να μειώσουμε τους άσκοπους υπολογισμούς.
Παρακάτω παραθέτουμε ένα script γραμμένο σε ’R’το οποίο προσπαθεί να δημιουργήσει έναν ορθογώνιο χώρο φάσεων. Εδώ, φαίνεται και ο τρόπος γραμμικοποίησης των παραμέτρων του uvspec. Δε θα περιγράψουμε τη λειτουργία του καθώς το ίδιο αποτέλεσμα μπορεί να επιτευχθεί με οποιαδήποτε γλώσσα προγραμματισμοί.
Αρχείο LBT_job_list_creationPBS.R
#!/usr/bin/env Rscript
#### Clear environment
#### ------------------------------------
closeAllConnections()
rm(list = (ls()[ls() != ""]))
Sys.setenv(TZ = "UTC")
tic <- Sys.time()
library(bitops)
## folder for this model family
## setwd('/mnt/lapmg_a/.../LibRadTranM/clear_H2O_LAP')
datafile <- "../clear_H2O_LAP.Rds"
outfilelist <- "jobs_args.list"
if (!file.exists(datafile)) {
stop("No previus results found")
}
#### Options to create multiple jobs
#### ----------------------
wvlngth_min <- 250
# wvlngth_max = 4000.0
wvlngth_max <- 5025
# spline_min = 250 spline_max = 4000 spline_stp = 1
altitude <- 62.694168/1000 ## for LAP 62.694168/1000.
number_of_streams <- 6
rte_solver <- "sdisort"
mol_modify_O3 <- seq(290, 370, 10) ## climatology or iterate
mol_modify_H2O <- seq(2, 37, 10) ## MM : kg/m^2
## Min sza for thessaloniki ~17.20. At sza=100
## something breaks !!!!
sza <- unique(
c(
seq(15, 95, 10),
seq(25, 90, 10),
0
)
)
atmosphere_file <- c("afglms", "afglmw")
source_solar <- c("kurudz_1.0nm", "kurudz_0.1nm", "kurudz_full")
albedo <- unique(
c(
seq(0.05, 0.15, 0.05),
0.07
)
)
#### create combination of all options given
#### --------------
todolisting <- expand.grid(
wvlngth_min = wvlngth_min, wvlngth_max = wvlngth_max,
atmosphere_file = atmosphere_file, source_solar = source_solar,
rte_solver = rte_solver, number_of_streams = number_of_streams,
sza = unique(sza),
mol_modify_O3 = unique(mol_modify_O3),
mol_modify_H2O = unique(mol_modify_H2O),
albedo = unique(albedo),
altitude = altitude, stringsAsFactors = FALSE
)
todolisting <- data.frame(todolisting, stringsAsFactors = FALSE)
colanmes <- names(todolisting)
#### load saved results
saved_results <- readRDS(datafile)
saved_results <- data.frame(saved_results, stringsAsFactors = FALSE)
saved_results$hostname <- as.character(saved_results$hostname)
### Find jobs to do
temptodo <- todolisting[, colanmes] ## will keep intact
tempsaved <- saved_results[, colanmes] ## used to compare
## combine with index
temptodo$coder <- "A"
tempsaved$coder <- "B"
temp <- rbind(tempsaved, temptodo)
uniqu <- !duplicated(temp[, colanmes])
## keep unique
temp <- temp[uniqu, ]
## dont include 'B' (saved results)
temp <- temp[temp$coder == "A", ]
## drop coder column
todolisting <- subset(temp, select = -coder)
if (length(todolisting[, 1]) <
1) stop("STOPED! No jobs to do")
## add job index to keep track
todolisting$job_id <- cksum(apply(todolisting, 1, paste, collapse = ""))
## creat file with list of jobs to submit serialize
cat("", file = outfilelist)
for (ri in 1:nrow(todolisting)) {
OptVect <- todolisting[ri, ]
cat(
sprintf(""),
sprintf("%s ", OptVect$job_id),
sprintf(
"atmosphere_file@@aattmmoo=%s.dat@", OptVect$atmosphere_file
),
sprintf(
"source@@solar@@ssoollaa=%s.dat@", OptVect$source_solar
),
sprintf("mol_modify@@O3@@%s@@DU@", OptVect$mol_modify_O3),
sprintf("mol_modify@@H2O@@%s@@MM@", OptVect$mol_modify_H2O),
sprintf("albedo@@%s@", OptVect$albedo),
sprintf("sza@@%s@", OptVect$sza),
sprintf("altitude@@%s@", OptVect$altitude),
sprintf("rte_solver@@%s@", OptVect$rte_solver),
sprintf("number_of_streams@@%s@", OptVect$number_of_streams),
sprintf(
"wavelength@@%s@@%s@", OptVect$wvlngth_min,
OptVect$wvlngth_max
),
sprintf("pseudospherical@"),
sprintf("quiet@"),
sprintf("\n"),
sep = "", file = outfilelist, append = TRUE
)
}
cat(
paste(
"Jobs to do: ", nrow(todolisting),
"\n"
)
)Διαχείριση αποτελεσμάτων.
Η αποθήκευση και χρήση των αποτελεσμάτων μπορεί να γίνει αρκετά δύσκολη μετά από κάποιο όγκο προσομοιώσεων. Όπως για παράδειγμα, όταν έχουμε να κάνουμε με διερεύνηση ατμοσφαιρικών συνθηκών, οπού πολύ εύκολα μπορούμε να φτάσουμε τα εκατομμύρια εκτελέσεων. Αυτό μπορεί να θέσει όρια λόγω της μνήμης του υπολογιστή, της πολυπλοκότητας αποθήκευση, του χρόνου αναζήτησης, του συστήματος αρχείων και άλλα. Για τον λόγο αυτό, προτείνεται η χρήση κάποιας βάσης δεδομένων του προτύπου SQL. Στην εφαρμογή μας χρησιμοποιήσαμε την βάση δεδομένων SQLite.
Επιγραμματικά αναφέρουμε κάποια οφέλη:
- Πρακτικά, απεριόριστη δυνατότητα αποθήκευσης σε ενιαίο αρχείο/πίνακα.
- Ταχύτατη αναζήτηση και ανάσυρση δεδομένων.
- Δυνατότητα απόρριψης διπλότυπων κατά την εισαγωγή δεδομένων.
- Συμβατή με τις περισσότερες γλώσσες προγραμματισμού και υπολογιστικών συστημάτων.
- Δυνατότητα αποθήκευση περιληπτικών αποτελεσμάτων και αντιστοίχηση με τα φασματικά.
Οδηγός Εγκατάστασης της LibRadtran σε περιβάλλον Windows.
Αρχικός οδηγός από τον Κώστα Φράγκο, προσαρμογή από Θανάση Νάτση.
Η LibRadtran είναι ένα πακέτο επίλυσης της εξίσωσης της διάδοσης της ακτινοβολίας μιας διάστασης (1D). Αποτελείται από ξεχωριστούς κώδικες γραμμένους σε γλώσσα ‘C’ ή/και ‘Fortran’. Πριν να είναι κάποιος σε θέση να χρησιμοποιήσει την LibRadtran θα πρέπει να “μεταγλωττίσει” και να “συνδέσει” (εγκατάσταση) τις ξεχωριστές ρουτίνες μεταξύ τους. Επειδή, το πακέτο είναι κατασκευασμένο να δουλεύει σε περιβάλλον Linux, για να το εγκαταστήσουμε σε περιβάλλον Windows απαιτούνται κάποιες επιπρόσθετες διαδικασίες, οι οποίες περιγράφονται βήμα-βήμα σε αυτόν τον οδηγό και περιγράφονται πιο συνοπτικά στις οδηγίες εγκατάστασης σε περιβάλλον Windows στη σελίδα της LibRadtran.
Εγκατάσταση του λειτουργικού περιβάλλοντος ‘Cygwin’.
Αρχικά εγκαθιστούμε τη σουίτα ‘Cygwin’, που μας επιτρέπει να εκτελούμε εντολές Linux. Κατά τη διάρκεια της εγκατάστασης του ‘Cygwin’ πρέπει να σιγουρευτούμε ότι έχουμε περιλάβει όλες τις απαραίτητες βιβλιοθήκες που απαιτούνται για την ομαλή εγκατάσταση της LibRadtran. Αυτές είναι οι ακόλουθες:
bash binutils cygwin flex gawk
gcc gzip make tarΑπό την ιστοσελίδα του Cygwin κατεβάζουμε το πρόγραμμα εγκατάστασης του Cygwin. Σημείωση ακόμα και αν έχουμε 64bit έκδοση λειτουργικού, καλό θα ήταν να εγκαταστήσουμε την 32bit έκδοση του ‘Cygwin’, για να αποφύγουμε στη συνέχεια κάποιες επιπλέον ρυθμίσεις.
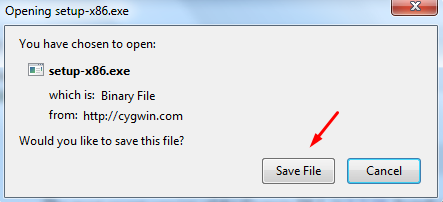
Σχήμα 1: Κατέβασμα και αποθήκευση του εκτελέσιμου προγράμματος εγκατάστασης του ‘Cygwin’.
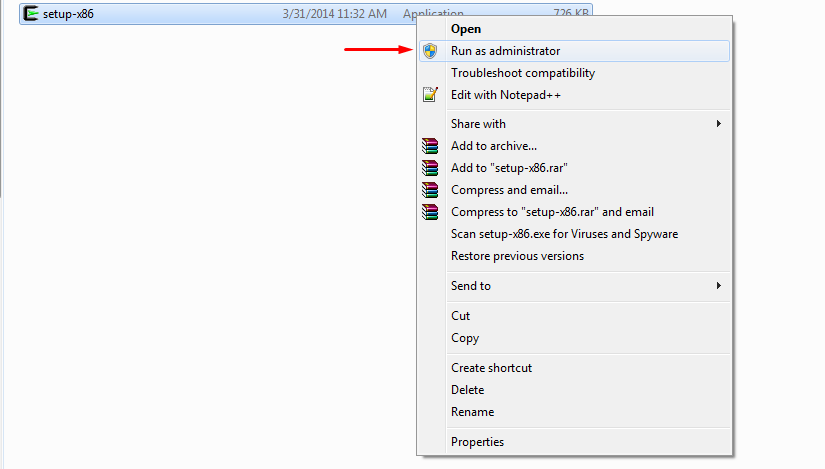
Αφού κατεβάσουμε το εκτελέσιμο (Εικόνα 1), πατάμε δεξί κλικ και επιλέγουμε ‘Run as Administrator’ (Εκτέλεση ως διαχειριστής) (Εικόνα 2).
Σχήμα 2: Εκτέλεση του προγράμματος εγκατάστασης του ‘Cygwin’.
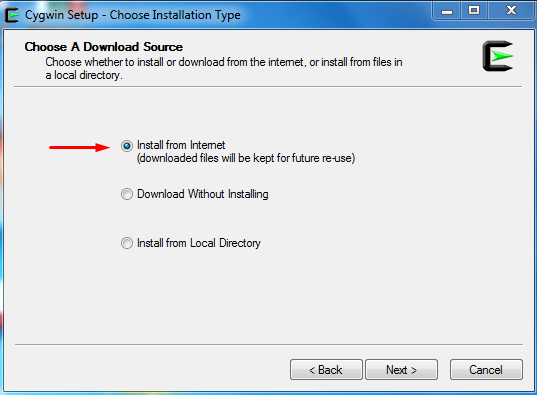
Στις επόμενες επιλογές πατάμε ‘yes’ και ‘Next’, μέχρι να δούμε την Εικόνα 3, όπου θα επιλέξουμε να κάνουμε εγκατάσταση από το διαδίκτυο (install from internet) και στη συνέχεια την επιλογή ‘Next’.
Σχήμα 3: Εγκατάσταση του Cygwin χρησιμοποιώντας το Internet.
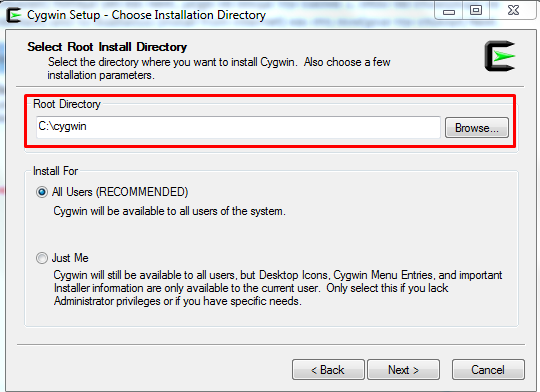
Επιλέγουμε να αφήσουμε το default directory εγκατάστασης (4) και συνεχίζουμε πατώντας ‘Next’. Είναι καλό ο φάκελος εγκατάστασης που θα επιλέξουμε να μην έχει κενά, να αποτελείται μόνο από λατινικούς χαρακτήρες και είναι δυνατόν να βρίσκεται στο c:\ (root των Windows).
Σχήμα 4: Επιλογή εγκατάστασης του Cygwin στο default directory.
Προσπερνάμε την επόμενη σελίδα που θα μας βγάλει, πατώντας ‘Next’ και στη συνέχεια επιλέγουμε τον τύπο σύνδεσής μας στο Internet (συνήθως είναι direct connection) και επιλέγουμε το ‘Next’ (Εικόνα 5).
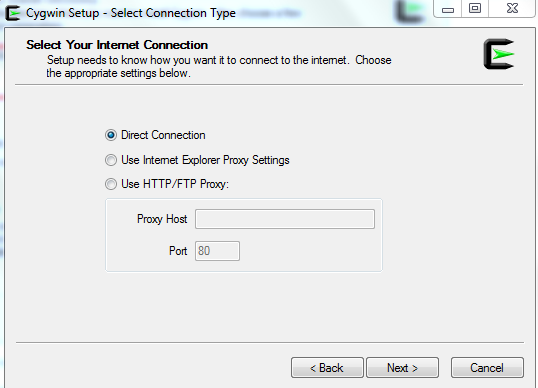
Σχήμα 5: Επιλογή σύνδεσης στο Internet.
Στην επόμενη καρτέλα διαλέγουμε από που θέλουμε να κατεβάσουμε τα πακέτα που είναι απαραίτητα για την εγκατάσταση του ‘Cygwin’. Κάνοντας scroll down με το ποντίκι μπορούμε να βρούμε κάποια τοποθεσία στην Ελλάδα (π.χ. στην Εικόνα 6 έχει γίνει επιλογή του ftp του Μετσόβιου Πολυτεχνείου. Κάποιος μπορεί να επιλέξει οποιαδήποτε τοποθεσία θέλει, επιλέγοντας κάποια τοποθεσία στην Ελλάδα ελαχιστοποιεί το χρόνο που χρειάζεται για να κατέβουν τα πακέτα εγκατάστασης).
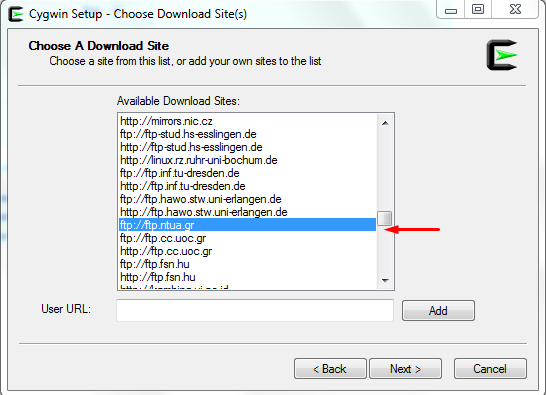
Σχήμα 6: Επιλογή τοποθεσίας για το κατέβασμα του ‘Cygwin’.
Στην επόμενη σελίδα θα διαλέξουμε τα πακέτα που χρειάζονται για την εγκατάσταση της LibRadtran τα όποια όπως είδαμε πιο πάνω είναι: bash, binutils, cygwin, flex, gawk, gcc, gzip, make, tar. Στο πλαίσιο που έχει για αναζήτηση (Εικόνα 7) τοποθετούμε καθένα πακέτο ξεχωριστά και κάνουμε αναζήτηση για να δούμε αν είναι προεγκατεστημένο ή χρειάζεται να το επιλέξουμε εμείς. Συνήθως, τα πακέτα βρίσκονται στις κατηγορίες ‘Base’, ‘Devel’ και ‘Utils’.
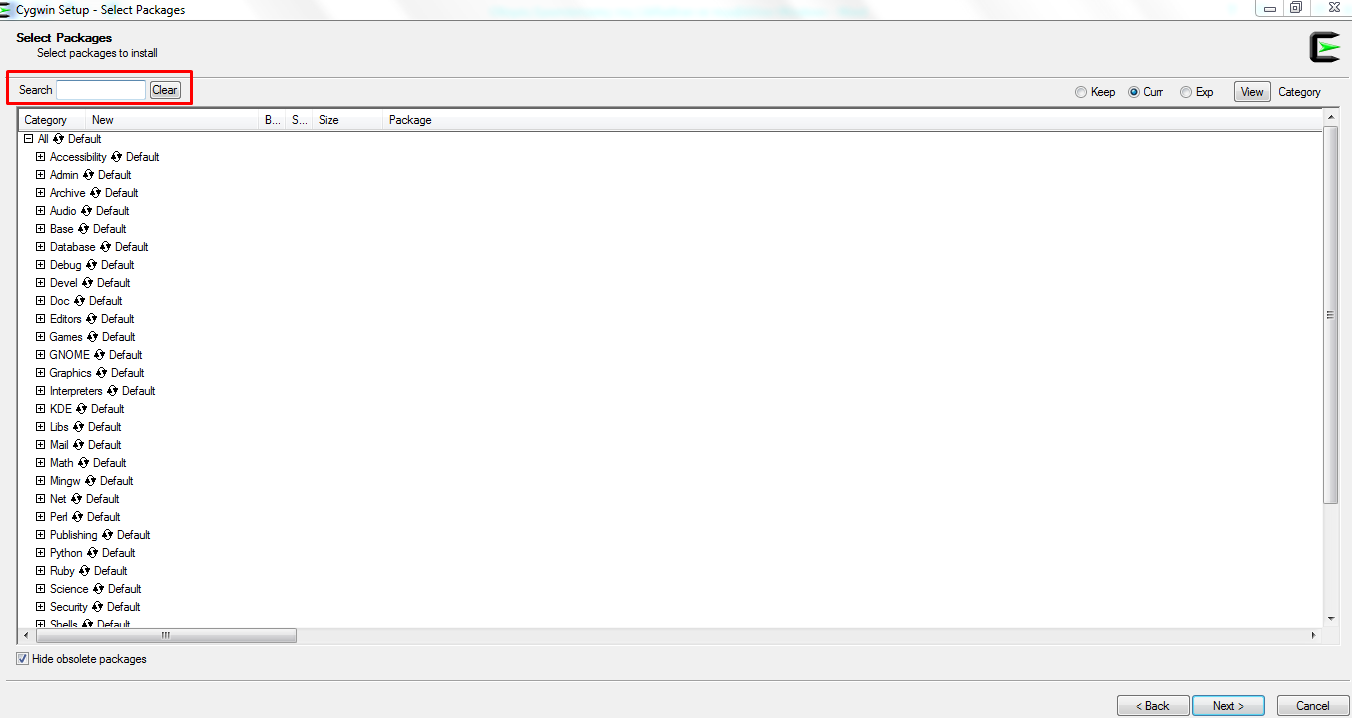
Σχήμα 7: Αναζήτηση των πακέτων που είναι απαραίτητα να εγκατασταθούν.
Ξεκινούμε αναζητώντας το πακέτο bash (Εικόνα 8) όπου παρατηρούμε ότι είναι ήδη στα πακέτα που γίνονται εγκατάσταση.
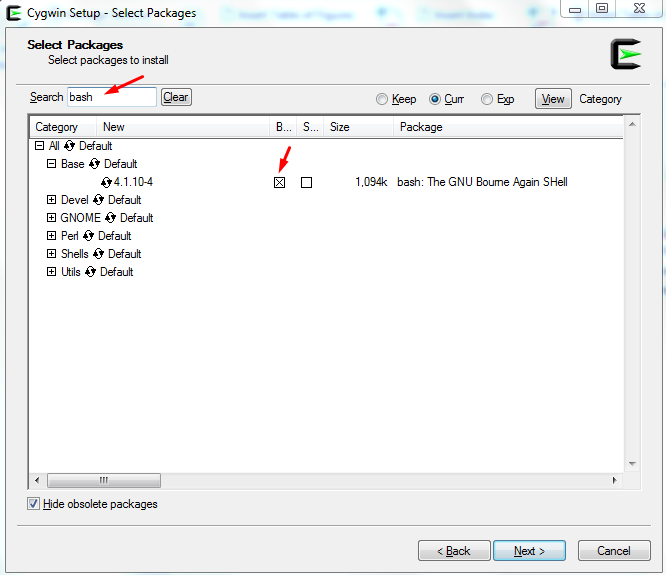
Σχήμα 8: Αναζήτηση του πακέτου bash, παρατηρούμε ότι είναι προεγκατεστημένα πακέτα του Cygwin.
Στη συνέχεια αναζητούμε το binutils, το οποίο είναι στο ‘Devel’ (Εικόνα 9) παρατηρούμε ότι έχει την επιλογή Skip, πατώντας μια φορά με το ποντίκι πάνω στο ‘Skip’, αλλάζουμε την κατάσταση ώστε να εγκατασταθεί, όπως φαίνεται στην Εικόνα 10. Την ίδια διαδικασία ακολουθούμε και για όλα τα υπόλοιπα πακέτα που παρατηρούμε ότι δεν περιέχονται από default (όπως το bash) στην εγκατάσταση του ‘Cygwin’.
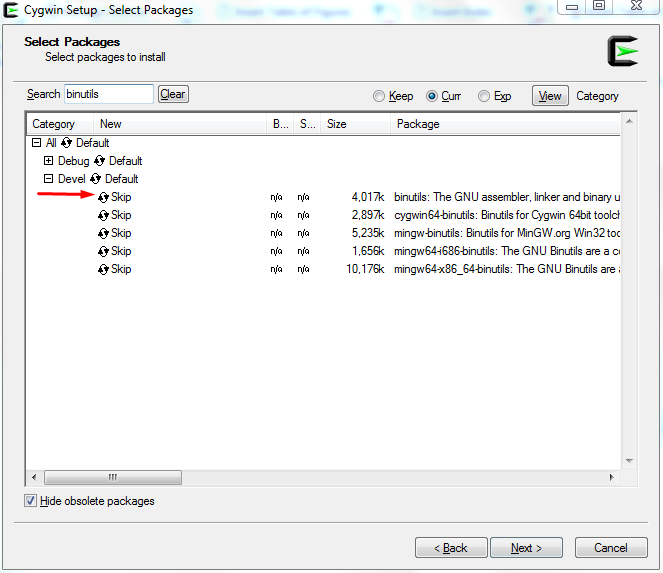
Σχήμα 9: Αναζήτηση της επιλογής binutils.
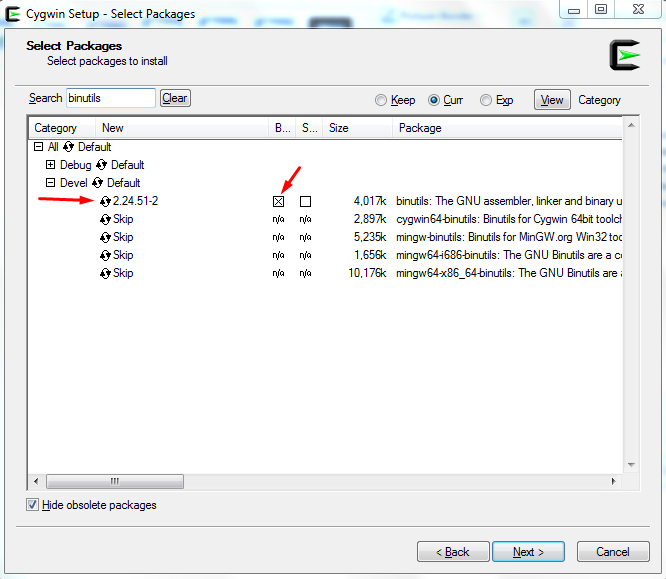
Σχήμα 10: Επιλογή εγκατάστασης του binutils.
Εν συνεχεία αναζητούμε το ‘Cygwin’, το οποίο όπως παρατηρούμε στην Εικόνα 11, εγκαθίσταται ως default επιλογή.
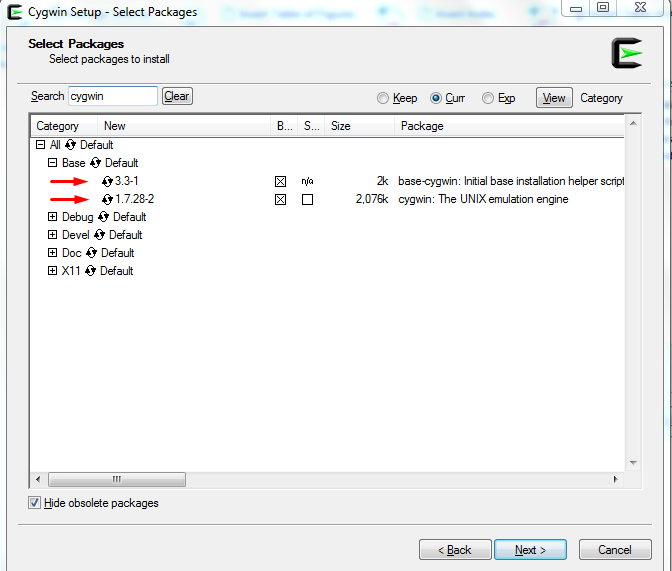
Σχήμα 11: Αναζήτηση του ‘Cygwin’, δε χρειάζεται κάποια μεταβολή.
Το πακέτο flex δεν περιέχεται στα default πακέτα εγκατάστασης, οπότε κάνουμε ότι για το πακέτο binutils, έτσι ώστε να δούμε την Εικόνα 12.
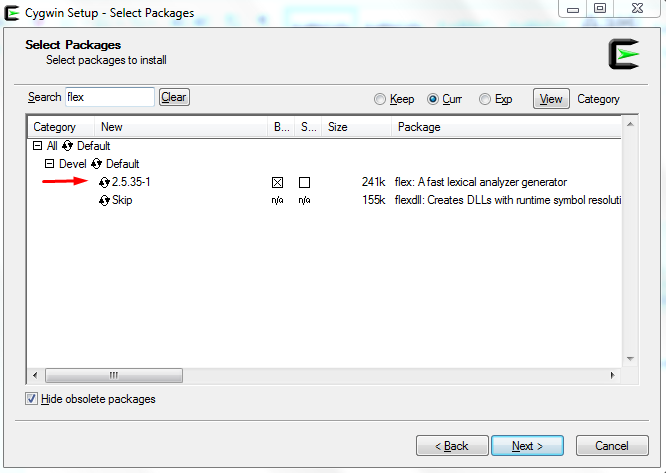
Σχήμα 12: Επιλογή εγκατάστασης του πακέτου flex.
Το πακέτο gawk, βρίσκεται ανάμεσα σε αυτά που εγκαθίστανται από default (Εικόνα 13).
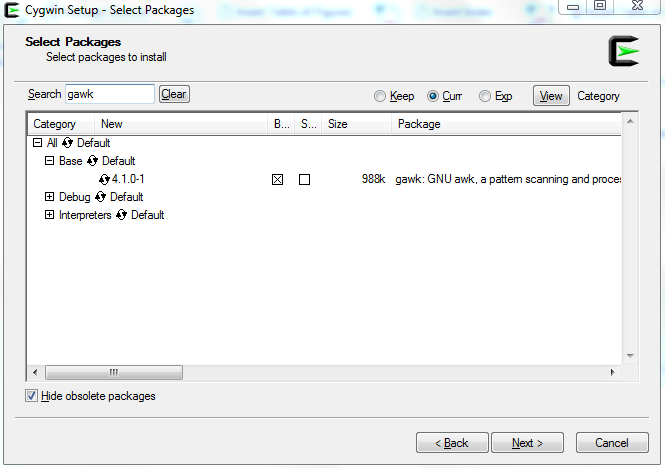
Σχήμα 13: Αναζήτηση του πακέτου gawk.
Ο gcc είναι ο compiler που μεταγλωττίζει κώδικα γλώσσας ‘C’ και ‘Fortran’. Επιλέγετε για εγκατάσταση αυτούς που φαίνονται στην Εικόνα 14.
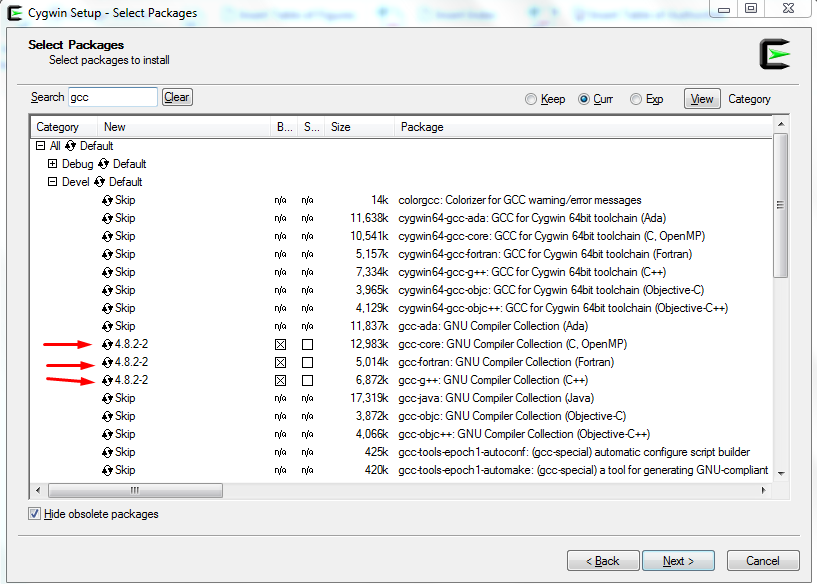
Σχήμα 14: Εγκατάσταση του compilers gcc για μεταγλώττιση του κώδικα C και Fortran.
To gzip είναι από τα default πακέτα του ‘Cygwin’ (Εικόνα 15).
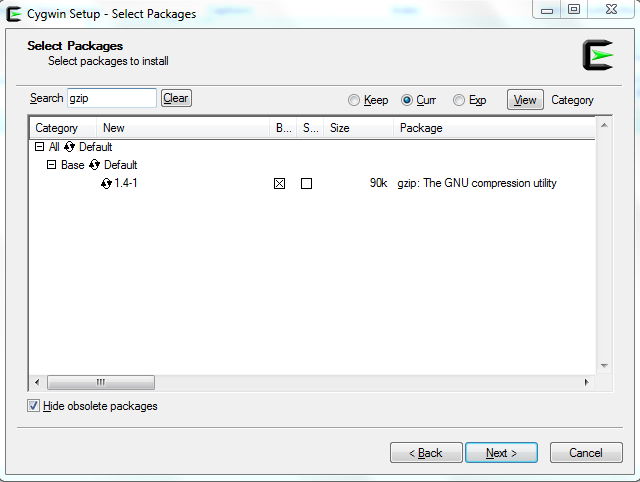
Σχήμα 15: Αναζήτηση του πακέτου gzip.
To make δεν είναι στα default οπότε χρειάζεται να αλλάξουμε την κατάστασή του για να εγκατασταθεί (Εικόνα 16).
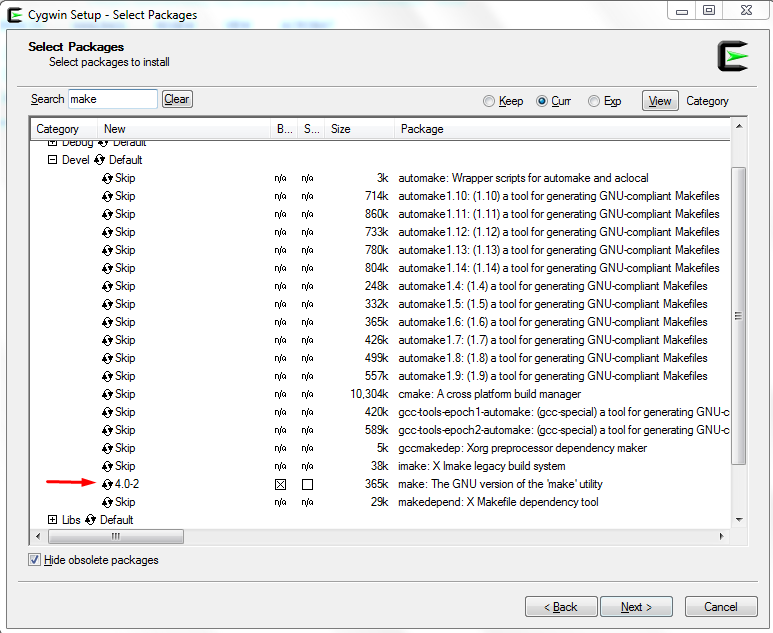
Σχήμα 16: Εγκατάσταση του πακέτου make.
To tar εγκαθίσταται από default (Εικόνα 17).
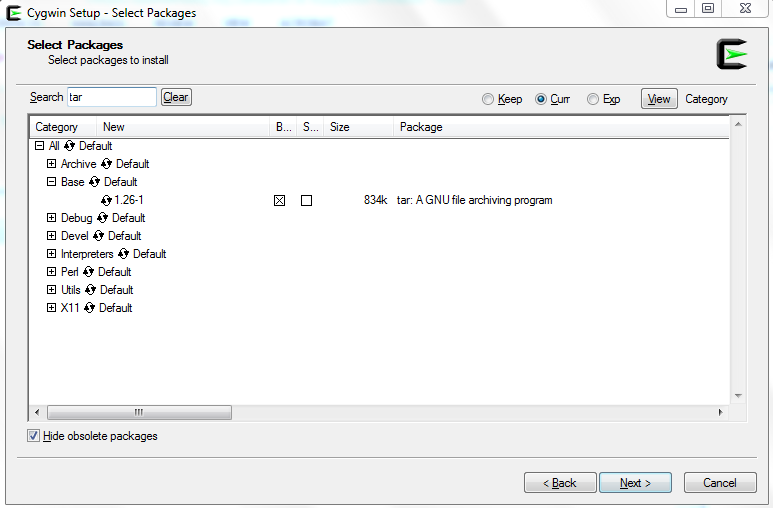
Σχήμα 17: Αναζήτηση του πακέτου tar.
Τέλος, εκτός από τα παραπάνω που είναι απαραίτητα για την εγκατάσταση της LibRadtran, καλό θα ήταν να εγκαταστήσουμε μαζί με το ‘Cygwin’ και τη γλώσσα ‘Perl’, αλλάζοντας το status από default σε install (Εικόνα 18).
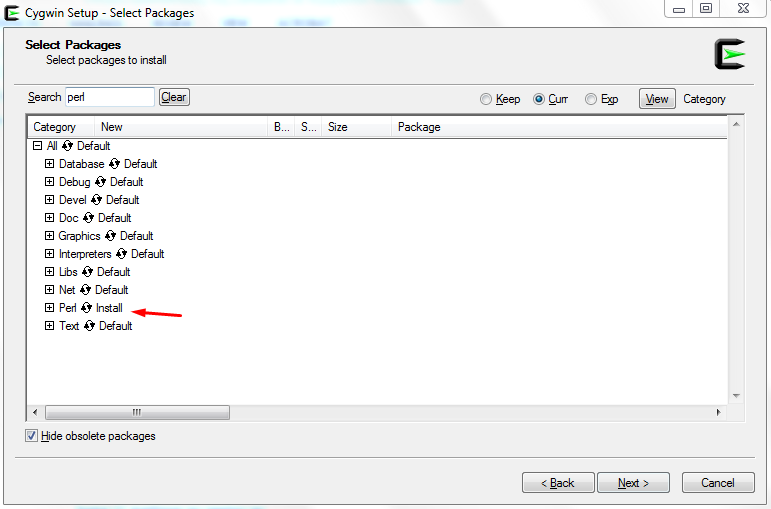
Σχήμα 18: Εγκατάσταση της γλώσσας ‘Perl’ σε περιβάλλον ‘Cygwin’.
Αφού ολοκληρώσουμε τον έλεγχο για τα πακέτα που θέλουμε να εγκαταστήσουμε πατάμε ‘Next’, όπου μετά θα δούμε όλα τα πακέτα που εγκαθίστανται και πατάμε ‘Next’, οπότε ξεκινάει το κατέβασμα και η εγκατάσταση του ‘Cygwin’ και των πακέτων που επιλέξαμε (Εικόνα 19).
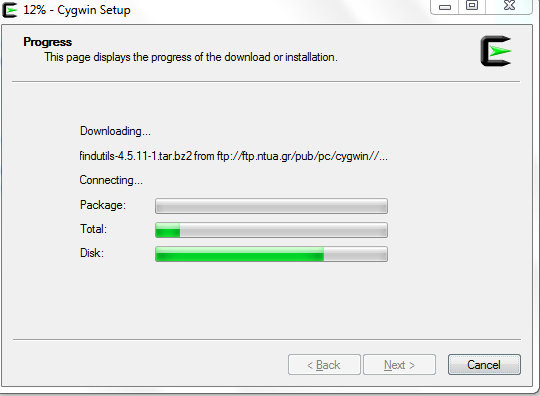
Σχήμα 19: Εγκατάσταση του ‘Cygwin’.
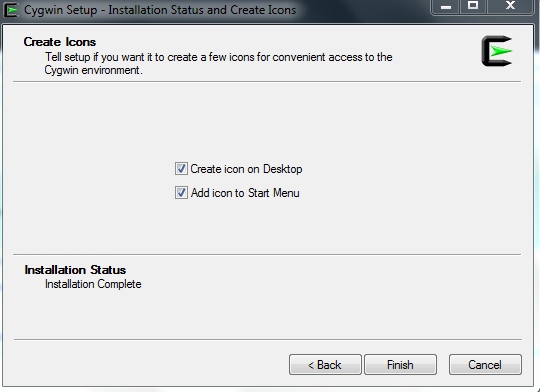
Σχήμα 20: Ολοκλήρωση εγκατάστασης του ‘Cygwin’.
Εγκατάσταση της LibRadtran.
Κατεβάζουμε την LibRadtran από το site. Παλιότερες εκδόσεις της LibRadtran μπορούν να βρεθούν εδώ.
Αφού ολοκληρωθεί η λήψη του αρχείου, το τοποθετούμε στο φάκελο c:\cygwin\home (Εικόνα 21).
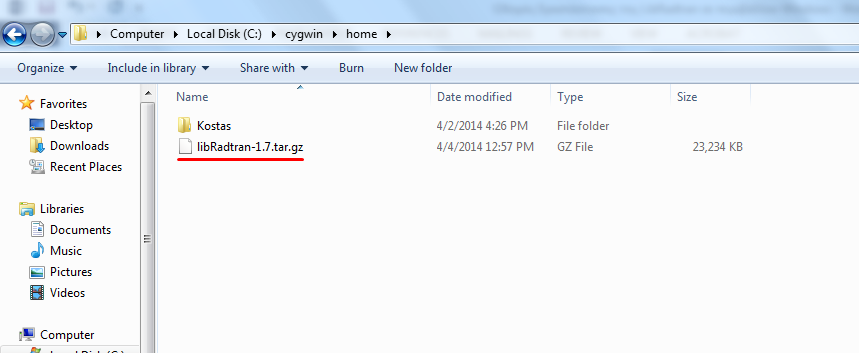
Σχήμα 21: Κατάλογος στον οποίο θα γίνει η εγκατάσταση της LibRadtran.
Ανοίγουμε το τερματικό του ‘Cygwin’ από την επιφάνεια εργασίας, εκτελούμε την εντολή cd .. για να βρεθούμε στο ‘home’ και στη συνέχεια την εντολή ls για να σιγουρευτούμε ότι έχουμε το πακέτο της libRadtran στον κατάλογο που βρισκόμαστε (Εικόνα 22).
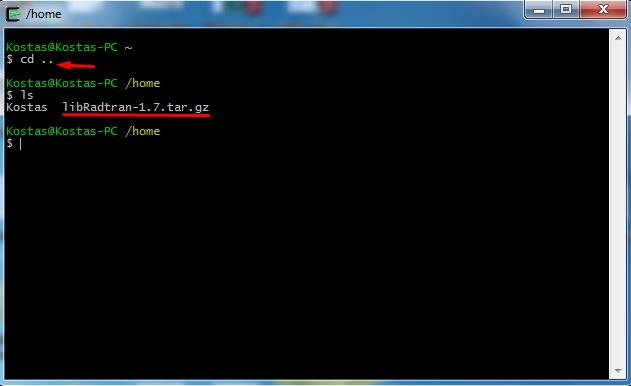
Σχήμα 22: Κατάλογος εργασίας στο περιβάλλον ‘Cygwin’.
Αφού σιγουρευτούμε ότι έχουμε το πακέτο της LibRadtran στον φάκελο που βρισκόμαστε εκτελούμε τις ακόλουθες εντολές, για την αποσυμπίεση των αρχείων.
gzip -d libRadtran-1.7.tar.gz
tar -xvf libRadtran-1.7.tarΣτην συνέχεια, με την εντολή ls επαληθεύουμε ότι έχει δημιουργηθεί ένας νέος φάκελος (Εικόνα 23).
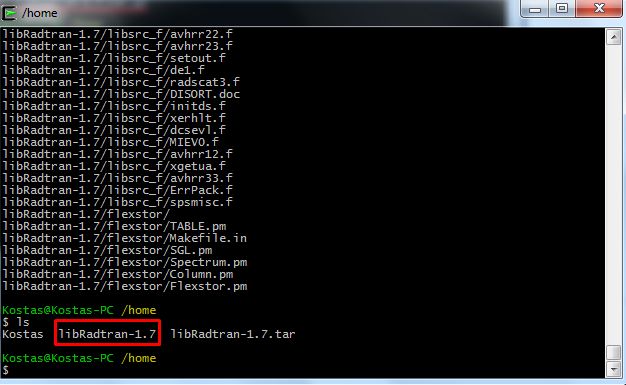
Σχήμα 23: Αποσυμπίεση του αρχείου Libradtran.tar.
Γράφουμε cd libRadtran-1.7, για να μεταφερθούμε στον φάκελο της Libradtran, με ls μπορούμε να δούμε τα περιεχόμενα του φακέλου και στη συνέχεια γράφουμε την ακόλουθη εντολή, για να επιλέξουμε τον compiler για την μεταγλώττιση του κώδικα ‘Fortran’ (Εικόνα 24).
export F77=gfortran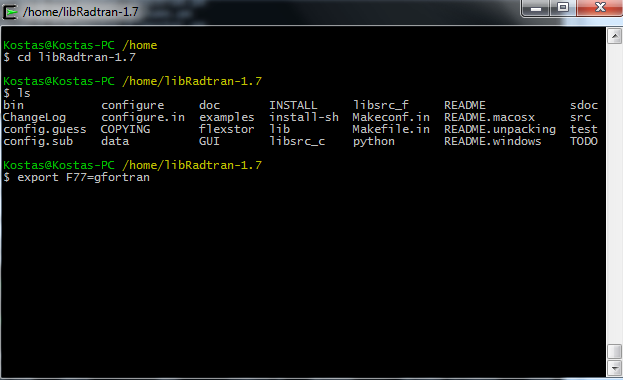
Σχήμα 24: Επιλογή του compiler για την αποσφαλμάτωση του κώδικα Fortran.
Τώρα εκτελούμε την εντολή ./configure. Στο τέλος, θα πρέπει να δούμε στην οθόνη του τερματικού τα ακόλουθα όπως φαίνεται στην Εικόνα 25.
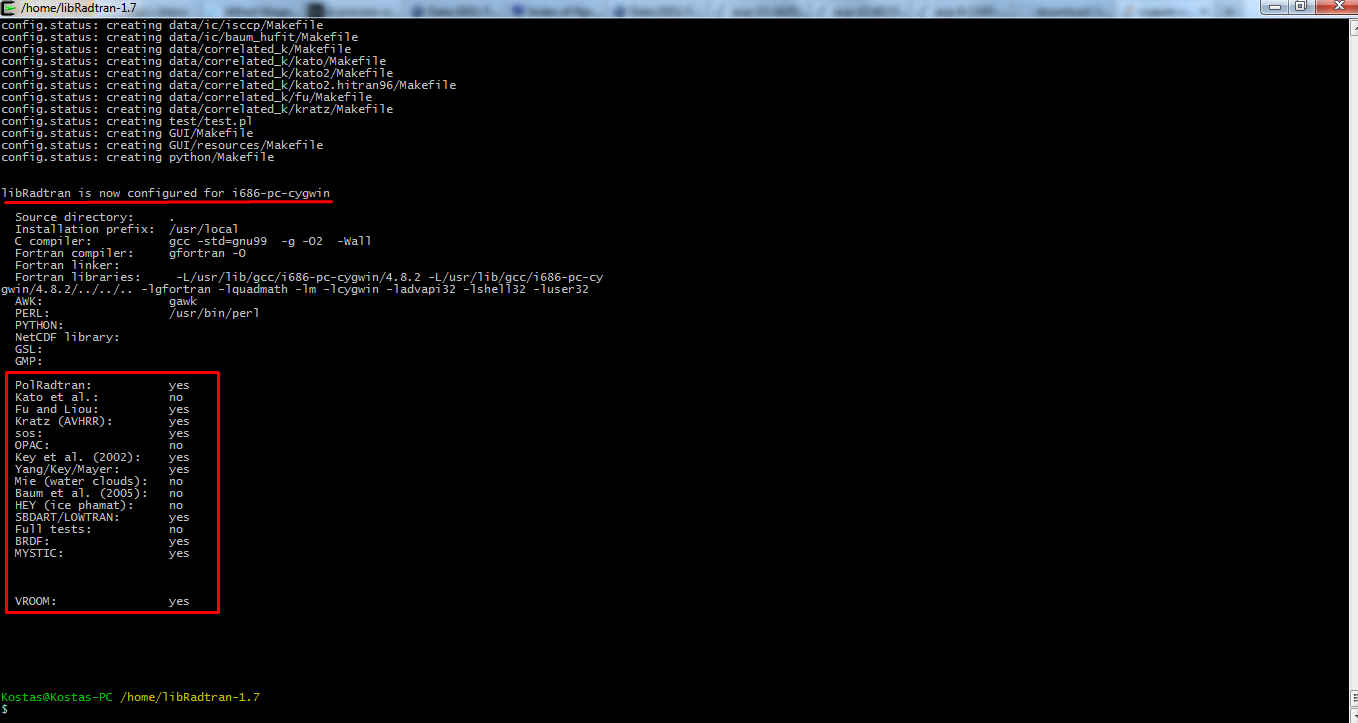
Σχήμα 25: Ολοκλήρωση της διαδικασίας configure.
Η εγκατάσταση (compile) τώρα μπορεί να ολοκληρωθεί εκτελώντας την εντολή make και για σιγουρευτούμε ότι όλα είναι εντάξει κατά τη διάρκεια της εγκατάστασης, αφού τελειώσει η εντολή make, εκτελούμε την εντολή make check, όπου θα δούμε στην οθόνη του τερματικού μας μια σειρά από test, όπως στην Εικόνα 26.
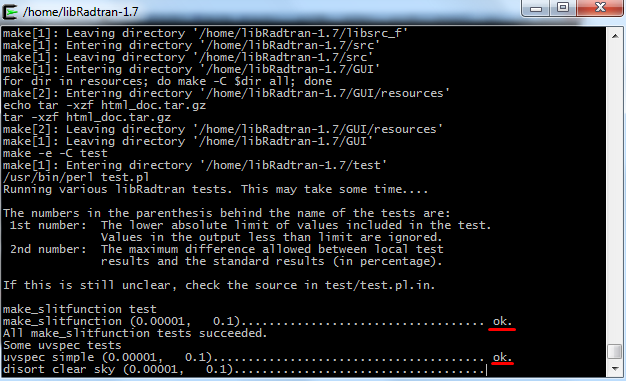
Σχήμα 26: Έλεγχος της ορθής εγκατάστασης της Libradtran.
Εάν όλα έχουν ολοκληρωθεί επιτυχώς, στον φάκελο libRadtran-1.7/bin υπάρχουν μια σειρά από εκτελέσιμα αρχεία. Για την χρήση τους μπορείτε να ανατρέξετε στο manual της libRadtran ή στα αντίστοιχα αρχεία του πηγαίου κώδικα (source code) που πολλές φορές, μπορεί να είναι πολύ πιο κατατοπιστικά για τη λειτουργία που εκτελούν. Το κύριο εκτελέσιμο αρχείο του μοντέλου είναι το uvspec.
Χρήσιμες πληροφορίες για τη χρήση της libRadtran.
Μονάδες μέτρησης ηλιακού φάσματος.
Οι μονάδες μέτρησης των αποτελεσμάτων είναι ίδιες με του παραμετρικού αρχείου εισόδου του ηλιακού φάσματος. Για παράδειγμα, για το αρχείο ‘kurudz_0.1nm.dat’ αναφέρεται ότι:
The original Kurudz [1992] data were converted to \(mW / (m^2 nm)\) and averaged over \(0.1nm\) intervals centered around the given wavelength.
Φασματικοί υπολογισμοί (spectral resolution).
Το βήμα των φασματικών υπολογισμών είναι προκαθορισμένο εσωτερικά. Για την αλλαγή του spectral resolution πρέπει να καθοριστεί μέσω της παραμέτρου wavelength_grid_file.
Έξοδος του uvspec.
Η τυπική διαμόρφωση (default output format) της εξόδου του uvspec είναι:
lambda edir edn eup uavgdir uavgdn uavgupΟι μεταβλητές εξόδου που μπορούν να δοθούν από το uvspec, ανάλογα με τα στοιχεία εισόδου, περιγράφονται παρακάτω.
- cmu
- Computational polar angles from polradtran.
- down_flux, up_flux
- The total (direct+diffuse) downward (down_flux) and up-ward(up_flux) irradiances. Same units as extraterrestrial irradiance ( e.g \(mW/(m^2 nm)\) if using the atlas3 spectrum in the data/solar_flux directory.)
- edir
- Direct beam irradiance w.r.t. horizontal plane (same unit as extraterrestrial irradiance).
- edn
- Diffuse down irradiance, i.e. total minus direct beam (same unit as edir).
- eup
- Diffuse up irradiance (same unit as edir).
- lambda
- Wavelength (\(nm\))
- u0u
- The azimuthally averaged intensity at numu user specified angles umu (units of e.g. \(mW/(m^2 nm sr)\) if using the atlas3 spectrum in the data/solar_flux directory.) Note that the intensity correction included in the disort solver is not applied to u0u, thus u0u can deviate from the azimuthally-averaged intensity-corrected uu.
- uavg
- The mean intensity. Proportional to the actinic flux: To obtain the actinic flux, multiply the mean intensity by \(4\pi\) (same unit as edir).
- uavgdir
- Direct beam contribution to the mean intensity (same unit as edir).
- uavgdn
- Diffuse downward radiation contribution to the mean intensity (same unit as edir).
- uavgup
- Diffuse upward radiation contribution to the mean intensity (same unit as edir).
- uu
- The radiance (intensity) at umu and phi user specified angles (unit e.g. \(mW/(m^2 nm sr)\) if using the atlas3 spectrum in the data/solar_flux directory.)
- uu_down, uu_up
- The downwelling and upwelling radiances (intensity) at cmu and phi angles (unit e.g. \(mW/(m^2 nm sr)\) if using the atlas3 spectrum in the data/solar_flux directory.)
Τυπικά ατμοσφαιρικά προφίλ.
Στον Πίνακα 1 παραθέτουμε στοιχεία από το ‘AFGL Atmospheric Constituent Profiles’ (Anderson et al. 1986).
| Model | Name | Lat | Time |
|---|---|---|---|
| 1 | Tropic | 15N | Annual Average |
| 2 | Mid-Latitude Summer | 45N | July |
| 3 | Mid-Latitude Winter | 45N | January |
| 4 | Sub Arctic Summer | 60N | July |
| 5 | Sub Arctic Winter | 60N | January |
| 6 | U.S. Standard | 1976 |
Προειδοποίηση για τη γωνία \({SZA}=43.2^\circ\).
H ζενίθια γωνία των \(43.2^\circ\) προκαλεί μία προειδοποίηση (warning). Το οποίο σχετίζεται με την δυνατότητα υπολογισμού τριγωνομετρικών συναρτήσεων μικρών γωνιών.
* * * * * * * WARNING > > > > > >
SETDIS--beam angle=computational angle;
* * * * * * * changing cosine of solar zenith angle, umu0, from 0.728969 to 0.728928